Progetto
Scopo di questo progetto è quello di integrare tre aree di ricerca: le proteine(proteomica), l’RNA attraverso i meccanismi di trascrizione (trascrittomica), e le mutazioni genetiche non attribuibili direttamente alla sequenza del DNA (epigenomica) in cervelli umani donati post-mortem a scopo di ricerca al fine di identificare le vie molecolari coinvolte nelle varie forme di demenza.
Progetto realizzato in collaborazione e con il sostegno paritetico di Fondazione Serpero e Fondazione Golgi Cenci, con la conduzione operativa di quest’ultima e il laboratorio di biologia dell’IRCCS Neurologico C. Mondino di Pavia.
RICERCA avviata a fine 2021, durata prevista 24 mesi
Obiettivi
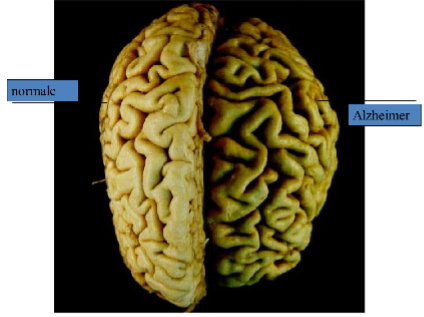
Questa ricerca vuole approfondire le caratteristiche biologiche presenti nelle diverse aree cerebrali che potrebbero aiutare a capire i meccanismi molecolari che favoriscono l’instaurarsi della demenza di Alzheimer e anche altre demenze. Con una stima di 50 milioni di persone colpite in tutto il mondo, le Demenze età-correlate hanno e avranno anche in futuro un peso economico e psicologico sulla nostra società [1], ma i meccanismi molecolari che predispongono o guidano tali patologie durante l’invecchiamento sono ancora oggi poco conosciuti. Per attuare queste analisi verranno impiegate le tecnologie high-throughput, per definire le firme “-omiche”, cioè un vasto ventaglio di molecole da poter confrontare. Questo può essere fatto in materiali biologici diversi, singole cellule, organismi modello (animali) oppure tessuto cerebrale umano ottenuto da pazienti post-mortem.
Quello che ci caratterizza è la possibilità di usare materiale umano post-mortem di casi ben seguiti e studiati in vita. In particolare, per queste valutazioni, meglio descritte dopo, sono stati considerati 3 soggetti con demenza di Alzheimer, 3 soggetti con sinucleinopatia Lewy Type e 3 soggetti controllo, cognitivamente sani. Le 5 aree cerebrali analizzate sono: lobo temporale, lobo parietale, sostanza nera, ippocampo e cingolo anteriore.
Per la ricerca si stanno attuando tre linee “operative”:
Prima: Caratterizzazione neuropatologica
Le diverse aree cerebrali considerate verranno analizzate sulla base di un protocollo pubblicato da Poloni et al. [2]. In breve, per valutare il grado di patologia Alzheimer verranno utilizzati appositi anticorpi verso la beta amiloide e la fosfoTAU, nonché la colorazione Gallyas, per le placche. Invece per la valutazione della presenza di Levy Bodies verrà utilizzato l’anticorpo per l’alfa-sinucleina. Infine, per evidenziare l’eventuale presenza concomitante di depositi TDP-43 si userà l’anticorpo anti-TDP-43.
Seconda: Analisi del trascrittoma
Mediante un approccio di RNA-Seq sarà possibile identificare sia gli RNA codificanti (RNA messaggero) sia quelli non codificanti (long non-coding RNA, microRNA, RNA circolari), per verificare quali tra questi sono deregolati nel contesto patologico e quali potrebbero diventare potenziali biomarcatori e/o essere utilizzati in nuovi approcci terapeutici[4,5].
Terza: Analisi epigenomica
Verrà studiata la metilazione globale del DNA. Le regioni del DNA differenzialmente metilate potranno essere utilizzate come indicatori dello stato di malattia. Mentre, mediante altra tecnologia, verranno studiate le modificazioni istoniche, in particolare metilazione e acetilazione [6,7].
Significato
Grazie all’utilizzo complementare della caratterizzazione trascrittomica ed epigenomica di varie aree cerebrali sarà possibile comprendere l’impatto e l’importanza dei cambiamenti nel panorama regolatorio del DNA sulle diverse tipologie di demenza. In particolare i dati generati da questo studio avranno l’enorme potenziale di consentire una migliore interpretazione funzionale delle varianti genetiche che risiedono in quelle aree cerebrali o di marcatori epigenetici associati alla malattia indipendentemente dalla genetica.
I risultati ottenuti in questo studio sono le premesse necessarie per permettere anche lo sviluppo di nuove terapie, in particolare di quelle epigenetiche che, rispetto alle terapie tradizionali, presentano diversi vantaggi.
L’utilizzo di possibili terapie epigenetiche si inserirebbe poi perfettamente nel contesto della Medicina Personalizzata: “La cura giusta, al momento giusto, per il paziente giusto” che è il fulcro del presente progetto. Questa terapia potrà essere:
- Personalizzata: si genera una mappa unica del cervello di soggetti affetti da Demenza di vario tipo e questo sarà fondamentale per definire mediante criteri rigorosi la patogenesi individuale, specifica di quel soggetto.
- Predittiva: la correlazione tra le caratteristiche cliniche e la mappa molecolare del cervello potrà fornire indicazioni sulla traiettoria della malattia e sulla prognosi.
- Preventiva: dalla previsione di una traiettoria di malattia si potranno ipotizzare strategie interventistiche mirate a diversi livelli di alterazione molecolare e strutturale.
- Partecipativa: i dati potranno essere estesi e valutati su altri donatori afferenti alla Banca del Cervello; infatti dalla mappatura molecolare potrebbero derivare informazioni utili per seguire e prevedere le traiettorie cognitive dei donatori durante il follow-up della Banca del Cervello.
La durata complessiva della ricerca sarà di 24 mesi
Bibliografia
- Alzheimer’s Disease International, 2015 [Internet]. [cited 19 Feb 2021]. Available: https:\/\/fondazioneserpero.it//fondazioneserpero.it//www.alzint.org/u/WorldAlzheimerReport2015.pdf
- Poloni TE, Medici V, Carlos AF, Davin A, Ceretti A, Cassini P, et al. Abbiategrasso Brain Bank Protocol for Collecting, Processing and Characterizing Aging Brains. JOVE. Available: https:\/\/fondazioneserpero.it//fondazioneserpero.it//www.jove.com/video/60296/abbiategrasso-brain-bank-protocol-for-collecting-processing
- Choudhary C, Mann M. Decoding signalling networks by mass spectrometry-based proteomics [Internet]. Nature Reviews Molecular Cell Biology. Nat Rev Mol Cell Biol; 2010. pp. 427–439. doi:10.1038/nrm2900
- Zucca S, Gagliardi S, Pandini C, Diamanti L, Bordoni M, Sproviero D, et al. RNA-seq profiling in peripheral blood mononuclear cells of amyotrophic lateral sclerosis patients and controls. Scientific Data. Nature Publishing Groups; 2019;6. doi:10.1038/sdata.2019.6
- Gagliardi S, Zucca S, Pandini C, Diamanti L, Bordoni M, Sproviero D, et al. Long non-coding and coding RNAs characterization in Peripheral Blood Mononuclear Cells and Spinal Cord from Amyotrophic Lateral Sclerosis patients. Scientific Reports. Nature Publishing Group; 2018;8. doi:10.1038/s41598-018-20679-5
- Roadmap Epigenomics Consortium, Kundaje A, Meuleman W, Ernst J, Bilenky M, Yen A, et al. Integrative analysis of 111 reference human epigenomes. Nature. Nature Publishing Group; 2015;518: 317–329. doi:10.1038/nature14248
- Horvath S. DNA methylation age of human tissues and cell types. Genome Biology. Genome Biol; 2013;14. doi:10.1186/gb-2013-14-10-r115
- Zhu J, Adli M, Zou JY, Verstappen G, Coyne M, Zhang X, et al. Genome-wide chromatin state transitions associated with developmental and environmental cues. Cell. Cell; 2013;152: 642–654. doi:10.1016/j.cell.2012.12.033